- PCA는 데이터의 숨겨진 패턴을 드러내줍니다.
- Feature Space의 데이터 분포 중 가장 넓게 분포한 것부터 찾아주기 때문에 X Feature들만으로는 보기 어려운 패턴을 찾을 수 있습니다.
- X 인자들의 분포 패턴에 Y feature를 얹어서 그려봅시다.
- Feature engineering을 위한 실마리를 찾고자 합니다.
1. 데이터
이번 예시는 현업 데이터를 그대로 가져왔습니다.
데이터 공개나 자세한 설명이 어려운 점 양해바랍니다.
X data: 22개의 인자, 250만 줄 이상.
Y data: 1개의 인자, 불균형 Regression 문제.
가장 큰 어려움 중 하나는 Y 데이터의 불균형이 극심하다는 점입니다.
Y > 0.1이 전체의 0.8%, Y > 0.5가 전체의 0.06%에 불과합니다.
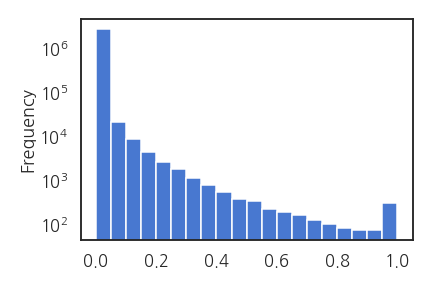
2. 전처리
2.1. outlier 제거
- 사전 작업을 통해 데이터의 outlier가 극심함을 알고 있습니다.
- 신뢰성있는 작업을 위해 outlier를 제거합니다.
- 대개 Q1과 Q3에서 IQR의 1.5배를 넘어서는 데이터를 outlier로 간주합니다.
- 정규분포에서 2.7$$\sigma$$에 해당하는 지점으로, 전체 데이터의 0.7% 가량이 outlier로 처리됩니다.
- 유사하게 데이터의 전체 비율 중 양 끝단을 제거하는 방식을 사용하겠습니다.
- 단, 제거 비율을 입력하면서 Y 데이터의 불균형에 미치는 영향을 확인하고, X 인자 중 0부터 시작하는 것은 이상치가 아니므로 제거하지 않습니다.
- 이상치 제거 함수를 작성합니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14def cut_quantile(x, lower, upper):
"""
x : (pd.Series)
lower : (float) lower bound to cut off
upper : (float) upper bound to cut off
"""
x_quantile = x.quantile([lower, upper])
lower_bound, upper_bound = x_quantile.values
if x.min() >= 0:
lower_bound = -np.finfo(float).eps
return x[(x > lower_bound) & (x < upper_bound)]
데이터의 극단으로부터 1% 이상과 0.1% 이상을 제거합니다.
1
2
3
4
5
6
7# 1% 제거
X_cut2 = X.apply(lambda x: cut_quantile(x, 0.01, 0.99), axis=0)
X_cut2.dropna(inplace=True)
# 0.1% 제거
X_cut3 = X.apply(lambda x: cut_quantile(x, 0.001, 0.999), axis=0)
X_cut3.dropna(inplace=True)확인 결과, 1% 제거시 전체의 16% 제거, 0.1%는 전체의 2.1%를 제거합니다.
의도와 달리 너무 많이 제거됩니다.
모든 인자가 극단에 있는 데이터가 드물다는 의미입니다.
X 이상치를 날린 후의 분포를 확인합니다. 시각화 코드는 생략합니다.
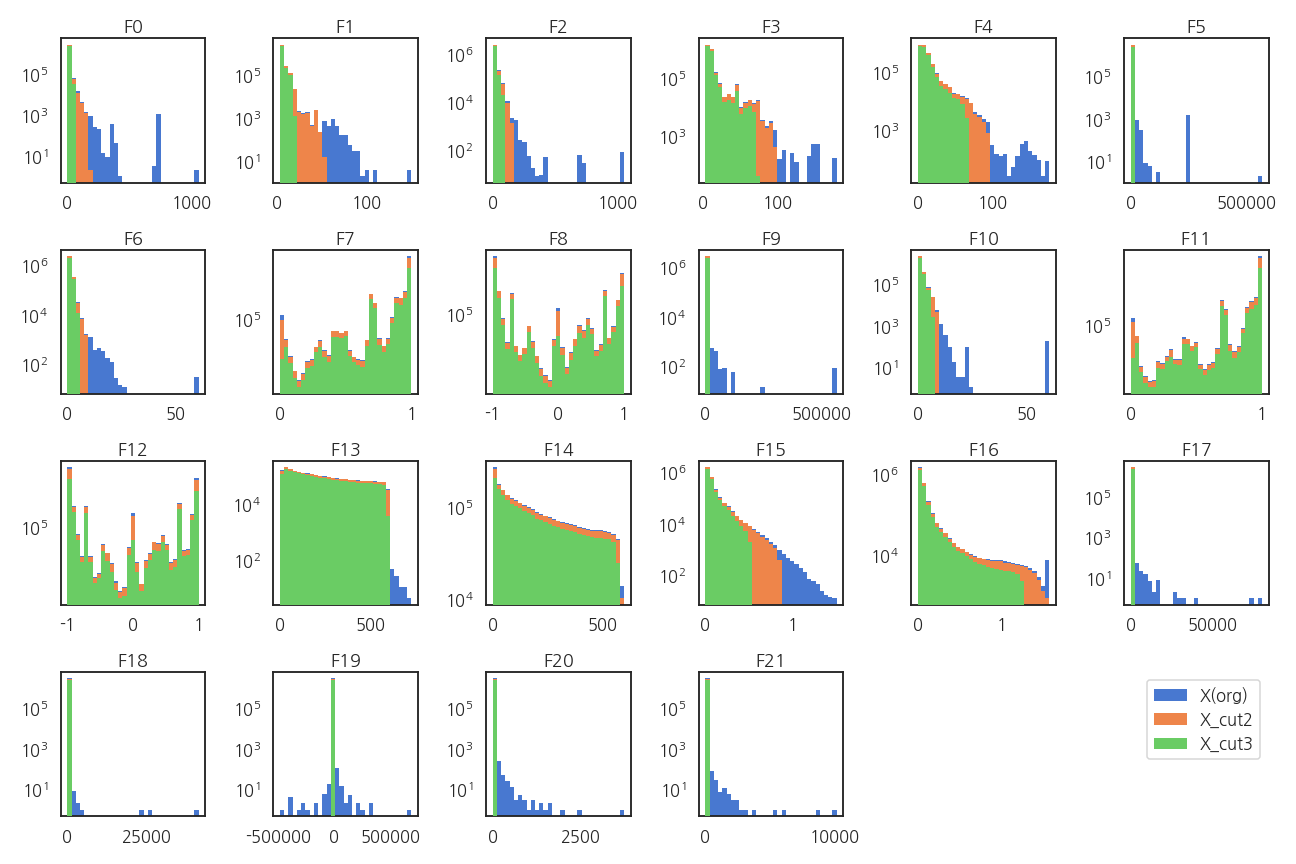
Y 인자는 무사한지 걱정이 됩니다.
X 이상치 제거로 Y 인자의 분포가 어떻게 변했는지 확인합니다.
히스토그램을 얻어서 구간별 데이터를 저장한 뒤 이상치 제거 전과의 차이를 봅니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28def plot_hist_delta(y1, y2, filename=None, bins=20):
fig, axs = plt.subplots(ncols=2, figsize=(10, 5))
counts_y1, bins_y1 = np.histogram(y1, bins=bins)
counts_y2, bins_y2 = np.histogram(y2, bins=bins)
axs[0].bar(bins_y1[:-1]+0.5*(bins_y1[1]-bins_y1[0]), counts_y1, width=(bins_y1[1]-bins_y1[0]))
axs[0].bar(bins_y2[:-1]+0.5*(bins_y2[1]-bins_y2[0]), counts_y2, width=(bins_y2[1]-bins_y2[0]))
axs[0].set_yscale("log")
axs[0].set_xlabel("Y")
axs[0].set_ylabel("counts (log)")
axs[1].bar(bins_y2[:-1]+0.5*(bins_y2[1]-bins_y2[0]),
1 - counts_y2/counts_y1,
width=(bins_y2[1]-bins_y2[0]),
fc="C2")
axs[1].set_ylim(0, 1.20)
axs[1].set_yticks(axs[1].get_yticks())
axs[1].set_yticklabels([f"{int(y*100)}%" for y in axs[1].get_yticks()])
axs[1].grid(axis="y")
axs[1].set_xlabel("Y")
axs[1].set_ylabel("$\Delta$ counts (log)")
fig.suptitle(filename)
fig.tight_layout()
if filename:
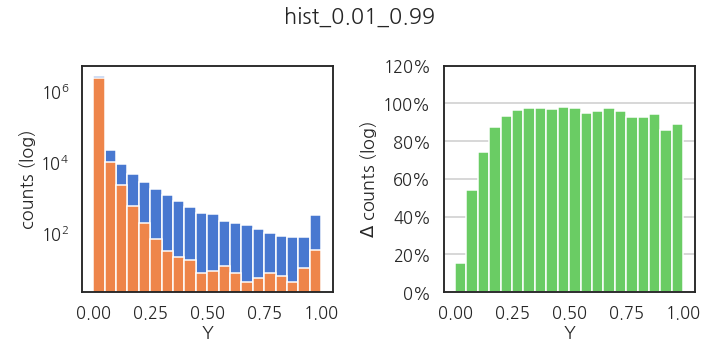
fig.savefig(f"./images/{filename}_post.png")1% 제거시: y 인자가 0 부근일 때를 제외하고 거의 다 날아갑니다.
1
plot_hist_delta(Y, Y_cut2, "hist_0.01_0.99")
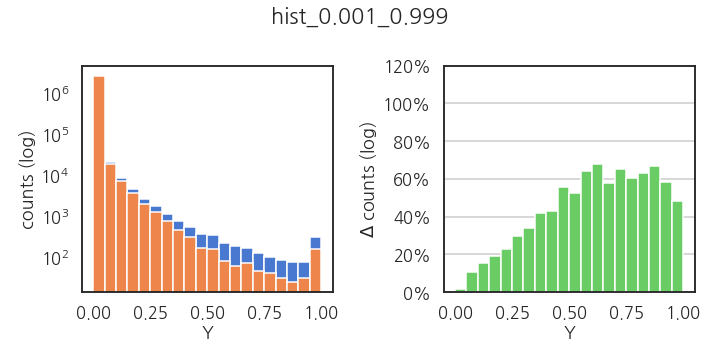
- 0.1% 제거시: 조금 낫지만 귀한 y > 0.5에서 손실률이 더 큽니다.
1
plot_hist_delta(Y, Y_cut2, "hist_0.001_0.999")
2.2. scaling
- PCA는 feature space의 분포를 기준으로 주성분을 결정합니다.
- 따라서 인자간 scaling이 매우 중요하며, 잘못되면 틀린 결론이 유도됩니다.
- 양 끝점을 0~1로 지정하는 MinMaxScaling을 수행합니다.
- 시험삼아 Std. Scaling을 섞어봤는데 무의미한 결과가 얻어지더군요.
1
2
3
4from sklearn import preprocessing
min_max_scaler = preprocessing.MinMaxScaler()
X_scaled_mm = pd.DataFrame(min_max_scaler.fit_transform(X))
3. PCA
3.1. explained variance ratio
- 이번 작업의 목적이 차원 축소는 아닙니다.
- 그러나 PCA가 효율적으로 수행되었는지 확인하기 위해 분산설명력을 확인합니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34def calc_evr(pca, filename=None):
# 1. explained variance ratio
pca_evr = pca.explained_variance_ratio_
print(f"# top 10 PCA explained ratio: {pca_evr[:10]}")
# 2. cumulative explained variance ratio
pca_cevr = np.array([0]+[pca_evr[:i].sum() for i in range(1, len(pca_evr))])
# 3. plot cumulative explained variance ratio
fig, ax = plt.subplots(figsize=(6, 4))
ax.plot(np.arange(len(pca_cevr)), pca_cevr, drawstyle="steps-post", c="b", marker="o", ms=7, mfc="w", mew=2, zorder=2)
ax.bar(np.arange(len(pca_cevr)+1), [0] + list(pca_evr), fc="gray", zorder=1)
xticks = [1, 5, 10, 15, 20]
xticklabels = [f"{int(x)}" for x in xticks]
ax.set_xticks(xticks)
ax.set_xticklabels(xticklabels)
ax.set_xlabel("Principle Component No.")
ax.set_ylabel("explained variance ratio")
ax.axhline(0.98, ls=":", c="gray")
ax.set_xlim(0.3,)
ax.set_ylim(0,)
ax.text(len(pca_cevr)-1, 0.85, "0.98", ha="right")
if filename:
fig.tight_layout()
filename_cevr = f"./images/PCA_normal_cevr_{filename}.png"
fig.savefig(filename_cevr)
return pca_evr, pca_cevr
pca = PCA()
X_pca = pca.fit_transform(X_scaled_mm)
pca_evr, pca_cevr = calc_evr(pca, filename="mm")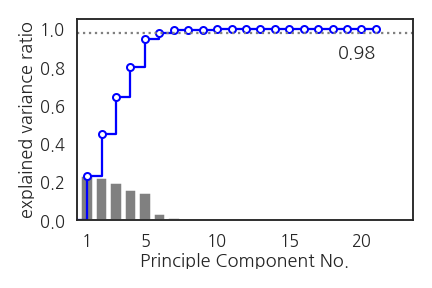
- 22개의 인자 중 처음 여섯 개만으로 98%의 분산이 설명됩니다.
- PCA가 상당히 효과적으로 진행되었음을 알 수 있습니다.
3.2. eigenvector
- PC1 ~ PC6까지 어떤 feature들로 구성되었는지 확인합니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22def calc_topn(pca, pca_cevr, filename="mm"):
# 1. get order of PCA, of which cumulative explained variance ratio > 0.95
PCn = np.where(pca_cevr > 0.98)[0][0]
print(f"PCn = {PCn}")
# 2. plot eigenvectors
fig, ax = plt.subplots(figsize=(9, 3), constrained_layout=True)
im = ax.imshow(pca.components_[:PCn], cmap="RdBu_r", vmin=-1, vmax=1)
ax.set_xticks(list(range(X.shape[1])))
ax.set_xticklabels([f"F{i}" for i, x in enumerate(X_cols)], rotation=90);
yticks = [int(i) for i in range(PCn)]
ax.set_yticks(yticks)
ax.set_yticklabels(f"PC{y+1}" for y in yticks)
fig.colorbar(im, ax=ax, fraction=0.04, aspect=20)
if filename:
filename_ev = f"./images/PCA_normal_ev_{filename}_post.png"
fig.savefig(filename_ev)
return PCn
PCn = calc_topn(pca, pca_cevr, filename="mm")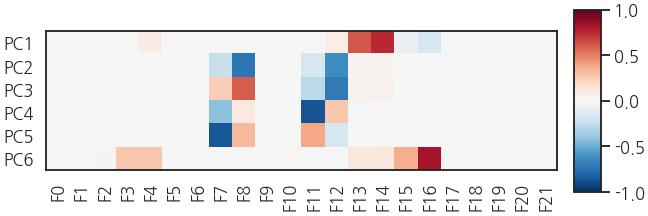
- 제법 흥미로운 결과가 나왔습니다.
- PC1과 PC2~PC5, PC6 세 개의 그룹으로 나눌 수 있는데 각 그룹에 사용된 인자가 독립적입니다.
- PC2~PC5도 언뜻 보기에 중구난방이지만 일종의 각도에 해당하는 인자이기 때문에 모두 물리적으로 의미가 있어보입니다.
- PC1~PC6까지 6차원 공간의 데이터 분포를 그리면 뭔가 보일 것 같습니다.
3.3. data distribution in PC space
PC 공간상의 데이터 분포를 보면 좋겠는데, 6차원은 2차원에 표현이 안됩니다.
6차원에서 두 개씩 뽑아서 2차원 그림 21개로 표현합니다.
데이터가 250만개가 넘습니다. 250만개 x 21개 > 5천만개를 넣으면 메모리가 걱정입니다.
작은 그림을 그려서 파일로 저장한 후 큰 그림에 붙여넣습니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32def plot_PCn_hist(X_pca, Y, PCn, filename=None):
Y_argsort = np.argsort(Y).values
if filename:
fig_all, axes_all = plt.subplots(ncols=PCn, nrows=PCn, figsize=(4*PCn, 4*PCn), sharex=True, sharey=True)
for i in range(PCn):
for j in range(1, PCn+1):
if i < j:
fig_s, ax_s = plt.subplots(figsize=(3, 3))
sns.histplot(x=X_pca[:,i][Y_argsort], y=X_pca[:,j][Y_argsort], bins=32, ax=ax_s)
ax_s.axhline(0, c="lightgray", zorder=0)
ax_s.axvline(0, c="lightgray", zorder=0)
ax_s.tick_params(axis="both", direction="in")
ax_s.set_xlabel(f"PC{i+1}")
ax_s.set_ylabel(f"PC{j+1}")
fig_s.tight_layout()
fig_s.savefig(f"./images/PCA_normal_{i+1}_{j+1}_hist.png");
im = plt.imread(f"./images/PCA_normal_{i+1}_{j+1}_hist.png")
axes_all[j-1, i].imshow(im);
axes_all[j-1, i].axis(False)
for i in range(0, PCn):
fig_all.align_ylabels(axes[:, i])
if filename:
fig_all.tight_layout()
fig_all.savefig(f"./images/PCA_normal_all_{filename}_hist.png")
plot_PCn_hist(X_pca, Y, PCn, filename="mm")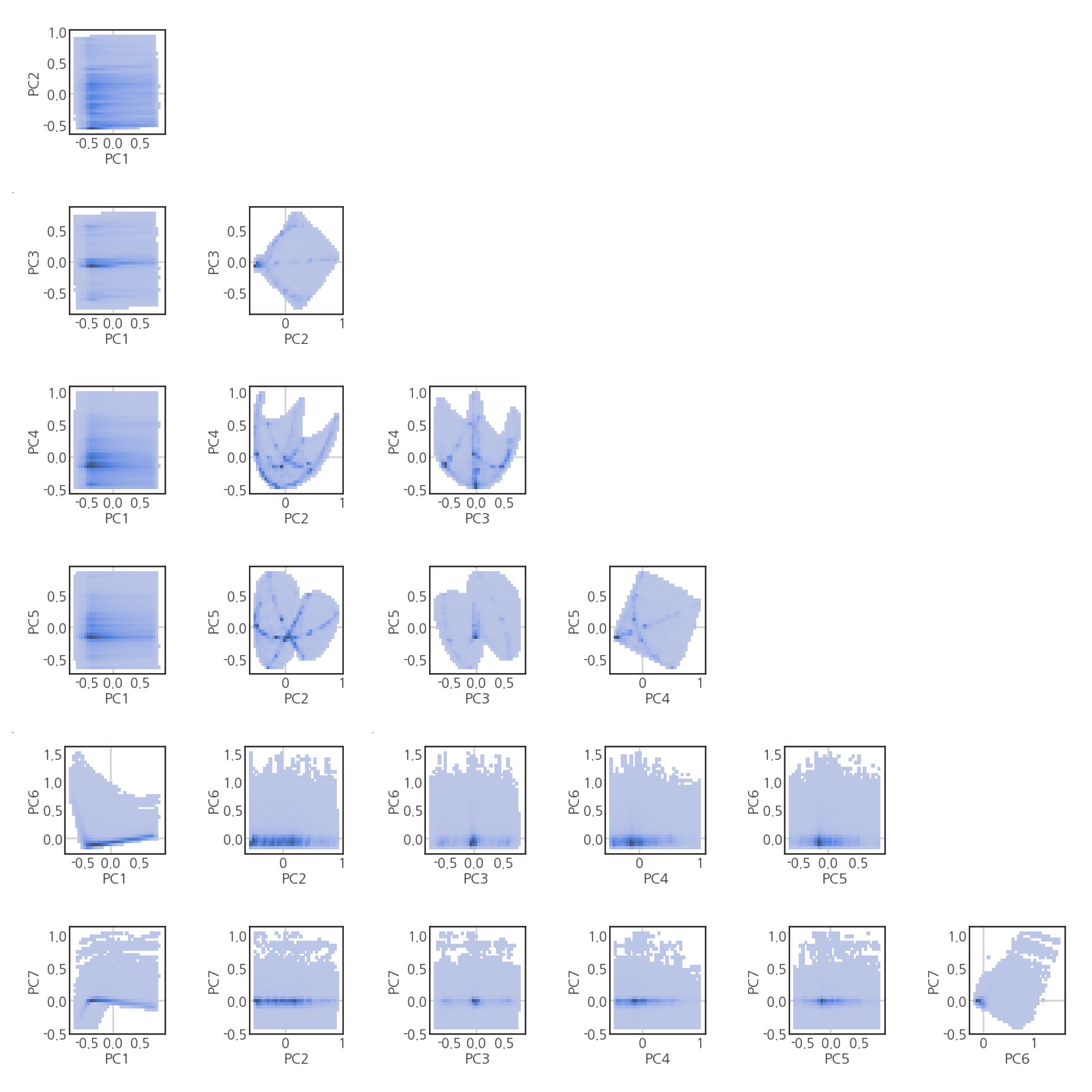
특정 지점의 데이터 밀도가 높습니다.
곳곳에 점이나 선 같은 패턴이 보입니다.
3.4. Y 인자 overlap
데이터가 어떻게 분포하는지 알았으니 Y 인자와는 어떻게 관련됐는지 보겠습니다.
scatter plot을 그리고 color에 Y 인자를 지정, colormap에 inferno를 지정합니다.
대부분의 데이터가 Y ~ 0이라서 그냥 그리면 큰 Y 값들이 묻혀 보이지 않습니다.
scatter plot을 그리기 전 데이터를 Y값 기준으로 정렬하면 보입니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32def plot_PCn(X_pca, Y, PCn, filename=None):
Y_argsort = np.argsort(Y).values
if filename:
fig_all, axes_all = plt.subplots(ncols=PCn, nrows=PCn, figsize=(4*PCn, 4*PCn), sharex=True, sharey=True, gridspec_kw={"hspace":0, "wspace":0})
for i in range(PCn):
for j in range(1, PCn+1):
if i < j:
fig_s, ax_s = plt.subplots(figsize=(3, 3))
ax_s.scatter(X_pca[:,i][Y_argsort], X_pca[:,j][Y_argsort], c=Y.values[Y_argsort], cmap="inferno", s=3, zorder=1)
ax_s.axhline(0, c="lightgray", zorder=0)
ax_s.axvline(0, c="lightgray", zorder=0)
ax_s.tick_params(axis="both", direction="in")
ax_s.set_xlabel(f"PC{i+1}")
ax_s.set_ylabel(f"PC{j+1}")
fig_s.tight_layout()
fig_s.savefig(f"./images/PCA_normal_{i+1}_{j+1}");
im = plt.imread(f"./images/PCA_normal_{i+1}_{j+1}.png")
axes_all[j-1, i].imshow(im);
axes_all[j-1, i].axis(False)
for i in range(0, PCn):
fig_all.align_ylabels(axes[:, i])
if filename:
fig_all.tight_layout()
fig_all.savefig(f"./images/PCA_normal_all_{filename}.png")
plot_PCn(X_pca, Y, PCn, filename="mm")
데이터 밀도가 높은 곳에 Y값이 큰 데이터가 놓여 있습니다.
패턴이 명확해서 살짝 들뜨면서도 이걸 어떻게 분류하나 싶어집니다.
3.5. 3D Plot
- 2차원 그림으로 두 변수 사이의 관계는 파악이 되지만, 여전히 저 그림들이 6D로 어떻게 맞물리는지는 머리가 좀 아픕니다.
- 제한적이나마 3D 그림을 그려봅니다.
- PC1, PC2, PC3를 골라서 3D Plot을 그려보겠습니다.
- 근데 3D 그림엔 한 가지 고려할 사항이 있습니다.
- 자연스러우려면, 멀리 있는 데이터가 가까이 있는 데이터에 가려져야 합니다.
- matplotlib은 시선의 방향을 파악하고 거기에 맞게 데이터를 정렬해 줍니다.
- interactive toy model로 확인합시다.
- jupyter notebook (lab)에 ipympl이 설치돼있다면
%matplotlib widget으로 interactive를 사용할 수 있습니다.1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17%matplotlib widget
X_tmp = np.array([[0.5, 0.5, -1], [0.5, -0.5, -1], [-0.5, 0.5, -1], [-0.5, -0.5, -1], [0.5, 0.5, 1], [0.5, -0.5, 1], [-0.5, 0.5, 1], [-0.5, -0.5, 1]])
elev = 30
azim = 40
fig = plt.figure(figsize=(5, 5))
ax = fig.add_subplot(111, projection='3d')
ax.scatter(X_tmp[:,0], X_tmp[:,1], X_tmp[:,2], ec="k", s=1500, alpha=1)
ax.view_init(elev=elev, azim=(azim-90))
ax.set_xlim(-2, 2)
ax.set_ylim(-2, 2)
ax.set_zlim(-2, 2)
ax.set_xlabel("X")
ax.set_ylabel("Y")
ax.set_zlabel("Z")
- 그런데 조금 억울합니다.
- 예전엔 시선에 자동으로 맞춰주는 기능이 없었거든요.
- 수동으로 데이터를 시선에 맞춰 정렬해줘야만 했습니다.
- 시선의 방향이 azimuthal angle, elevation(polar angle)으로 지정되어 있습니다.
- 내적을 통해 시선의 방향에 가까운 순으로 데이터를 정렬합니다.
- 확인을 위해 시선의 방향에 가까운 순으로 colormap을 입힙니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36%matplotlib inline
def calc_view(elev, azim):
return np.array([np.cos(np.deg2rad(azim-90))*np.cos(np.deg2rad(elev)), np.sin(np.deg2rad(azim-90))*np.cos(np.deg2rad(elev)), np.sin(np.deg2rad(elev))])
def sort_view(X, view):
return np.dot(X, view)
elev = 30
azim = -40
fig = plt.figure(figsize=(8, 4))
for i in range(2):
ax = fig.add_subplot(1, 2, i+1, projection="3d")
if i == 0: # not specified
ax.scatter(X_tmp[:,0], X_tmp[:,1], X_tmp[:,2],
ec="k", s=1500, alpha=1)
ax.set_title("default")
else: # specified
ax.scatter(X_tmp[:,0][viewsort], X_tmp[:,1][viewsort], X_tmp[:,2][viewsort],
c=sort_view(X_tmp, view), cmap="inferno",
ec="k", s=1500, alpha=1)
ax.set_title("specified")
ax.view_init(elev=elev, azim=(azim-90))
ax.set_xlim(-2, 2)
ax.set_ylim(-2, 2)
ax.set_zlim(-2, 2)
ax.set_xlabel("X")
ax.set_ylabel("Y")
ax.set_zlabel("Z")
fig.tight_layout()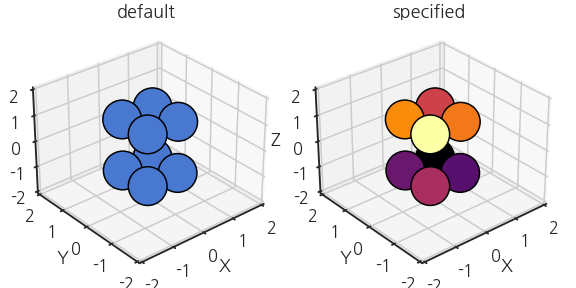
- 본 목적으로 돌아옵니다.
- PC1, PC2, PC3를 각기 x, y, z축으로 scatter plot을 그립니다.
- Y 인자값을 색상으로 입히는데, 앞에 있는 데이터에 뒤의 데이터가 가립니다.
- Y값이 큰 데이터의 위치를 파악하기 위해 $$Y > threshold$$를 적용하여 조금씩 그립니다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29fig = plt.figure(figsize=(20, 5))
for i, th in enumerate([0.8, 0.5, 0.3, 0.01], 1):
# data with Y > threshold
idx_3 = Y.loc[Y > th].index.values
# viewpoint
elev = 45
azim = -45
# sort data by viewpoint
view = calc_view(elev, azim)
idx_viewsort = np.argsort(sort_view(X_pca[idx_3,:3], view))
Y_viewsort = Y.loc[idx_3].values[idx_viewsort]
X_viewsort = X_pca[idx_3, :3][idx_viewsort]
ax = fig.add_subplot(1, 4, i, projection='3d')
ax.scatter(X_viewsort[:,0], X_viewsort[:,1], X_viewsort[:,2], c=Y_viewsort, cmap="inferno", s=20, vmin=0, vmax=1, alpha=1, lw=0.2, ec="k")
ax.view_init(elev=elev, azim=(azim-90))
ax.set_xlabel("PC1")
ax.set_ylabel("PC2")
ax.set_zlabel("PC3")
title = f"{th}"
if i == 1:
title = "threshold = "+ title
ax.set_title(title)
fig.tight_layout()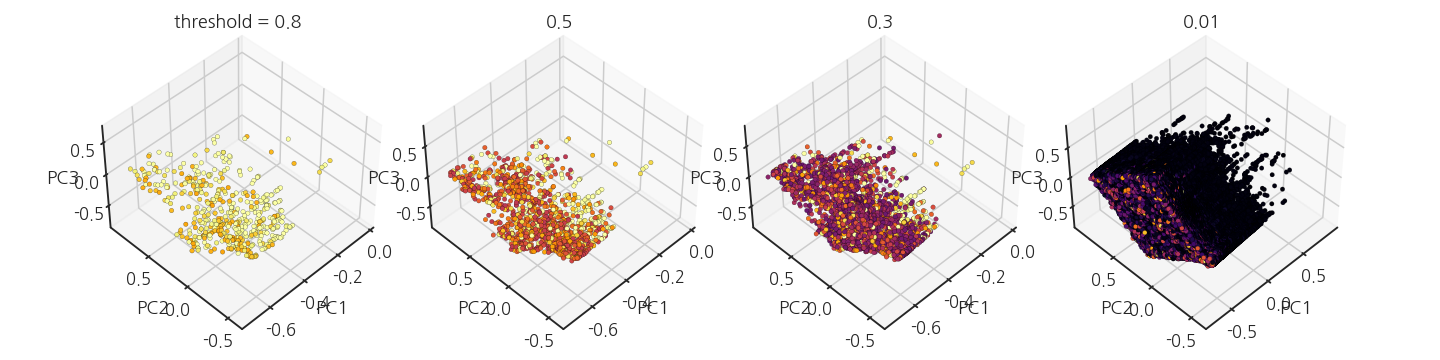
4. 결론
- PCA 결과를 가능한 그림으로 표현했습니다.
- X인자로 수행한 PCA의 데이터 분포를 파악하고, Y값과의 연관성을 파악했습니다.
- 데이터를 추가로 분석할 아이디어가 조금 생겼습니다.
- 좋은 결과로 이어지면 좋겠습니다.
